Что делать, если признаков в датасете слишком много для обучения модели Machine Learning (в том числе в Spark)? Один из вариантов — уменьшить размерность. Сегодня мы расскажем о доступном методе уменьшения размерности в Spark — PCA (Метод главных компонент). Читайте в этой статье: что такое PCA и зачем он нужен, к какому подходу выбора признаков он относится, инициализации PCA в Spark, а также применение метода уменьшения размерности на реальном датасете.
Что такое метод главных компонент (PCA)
PCA (Principal Component Analysis, Метод главных компонент) — это статистическая процедура уменьшения размерности. С математической точки зрения в основе PCA лежит нахождение линейно некоррелированных переменных, называемых главными компонентами. PCA очень часто используется в статистике и машинном обучении (Machine Learning), поскольку помогает выделить наиболее значимую информацию из всего датасета, который может состоять из сотни признаков.
Уменьшение размерности на основе метода главных компонент состоит из нескольких шагов:
- Данные выражаются в виде ортонормированных векторов, т.е. угол между ними равен 90°. Это реализуется за счет вычисления собственных векторов (eigenvectors).
- Эти векторы сортируются в порядке важности путем рассмотрения вклада каждого в разброс данных в целом.
- Выбираются только самые важные компоненты, т.е. те, которые объясняют данные более полно чем остальные. А поскольку они уже сортированы, то выбираются первые.
- Данные проецируются на выбранные компоненты.
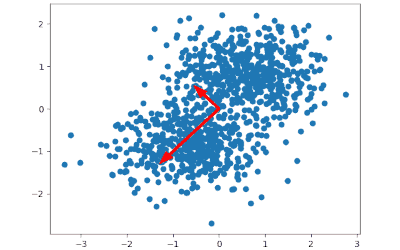
Зачем нужен метод главных компонент
Как правило, датасеты состоят из многих атрибутов. Когда этих атрибутов слишком много, то возникает проблема — какие из них выбрать для обучения модели Machine Learning, а может и вовсе взять все? При недостатке данных может возникнуть неодообучение, а при избытке — переобучение. А модель машинного обучения должна обобщать данные, а не подстраиваться под них. Мы не можем увеличивать количество признаков до бесконечности, но мы можем уменьшить размерность данных, например, с помощью PCA.
Есть два подхода к задаче отбора признаков:
- Feature Selection — процесс выбора подмножества соответствующих характеристик, которые используются для построении модели. В этом подходе измеряется вклад каждого признака в отдельности и в совокупности. Feature Selection может быть хорошей идеей, если предполагается, что большая часть отклонений в наборе данных объясняется несколькими переменными. Например, в прогнозировании цены на квартиру количество комнат является более важным признаком, чем цвет обоев.
- Feature Extraction начинается с начального набора данных и строит производные значения (характеристики), которые должны быть информативными и неизбыточными. Другими словами, будет создан производный набор данных, который можно будет использовать для обучения модели машинного обучения. Этот подход и реализуется за счет уменьшения размерности, в том числе PCA.
Feature Extraction предпочтительнее в том случае, когда каждое измерение вносит равный вклад. Также стоит выбрать Feature Extraction, если вы не имеете представления о вкладе каждой переменной в предсказательную силу модели (хотя если есть возможность, то лучше проверить).
Есть и другие методы уменьшения размерности, например, t-SNE, UMAP и др. Но в Spark реализован только PCA. Но не стоит расстраивается, поскольку PCA остается достаточно мощным инструментом и до сих пор используется в Machine Learning.
Как PCA работает в Spark
В Spark для инициализации метода главных компонент используется класс PCA. В аргументе нужно указать сколько компонент нужно извлечь. В примере ниже показано, как в Spark проецировать 5-мерные векторы признаков на 2-х мерные главные компоненты. Полученный столбец имеет название pcaFeatures. Как видите, в Spark метод главных компонент (PCA) реализуется несколькими строчками.
from pyspark.ml.feature import PCA
from pyspark.ml.linalg import Vectors
df = spark.createDataFrame(
data=[
(Vectors.dense([2.0, 0.0, 3.0, 4.0, 5.0]),),
(Vectors.dense([4.0, 0.0, 0.0, 6.0, 7.0]),),
(Vectors.dense([8.0, 1.0, 4.0, 9.0, 0.0]),),
],
schema=["features"]
)
pca = PCA(k=2, inputCol="features", outputCol="pcaFeatures")
model = pca.fit(df)
result = model.transform(df)
>>> result.show(truncate=False) +---------------------+---------------+ |features |pcaFeatures | +---------------------+---------------+ |[2.0,0.0,3.0,4.0,5.0]|[-0.357,2.855] | |[4.0,0.0,0.0,6.0,7.0]|[-0.157,7.433] | |[8.0,1.0,4.0,9.0,0.0]|[-9.435,5.219] | +---------------------+-------------- +
Пример использования PCA на реальном датасете
Воспользуемся классическим датасетом «Ирисы Фишера». Датасет разделен на три вида ириса по 50 экземпляров на каждый. Каждый экземпляр имеет 4 атрибута — длина/ширина наружной/внутренней доли околоцветника.
В Python-библиотеке Scikit-learn для загрузки датасета требуется лишь написать следующее:
from sklearn import datasets iris = datasets.load_iris() X = iris.data Y = iris.target # вид ириса, всего их 3 и пронумерованы (0, 1 или 2)
Данные хранятся в виде массивов NumPy, поэтому для создания Spark-датафрейма нужно преобразовать их в список. Пример кода для создания DataFrame в Spark на основе полученного датасета:
attributes = [
'Sepal Length',
'Sepal Width',
'Petal Length',
'Petal Width'
]
data = spark.createDataFrame(
data=X.tolist(),
schema=attributes)
>>> data.show(5) +------------+-----------+------------+-----------+ |Sepal Length|Sepal Width|Petal Length|Petal Width| +------------+-----------+------------+-----------+ | 5.1| 3.5| 1.4| 0.2| | 4.9| 3.0| 1.4| 0.2| | 4.7| 3.2| 1.3| 0.2| | 4.6| 3.1| 1.5| 0.2| | 5.0| 3.6| 1.4| 0.2| +------------+-----------+------------+-----------+
Поскольку алгоритмы Spark (в том числе PCA) требуют, чтобы данные были в виде одного вектора, а не разделены по разным атрибутам, то мы их преобразуем в один в с помощью VectorAssembler (о нем тут):
from pyspark.ml.feature import VectorAssembler
assembler = VectorAssembler(
inputCols=attributes,
outputCol="features"
)
output = assembler.transform(data)
Теперь можем применить метод главных компонент в Spark, как мы это делали выше:
from pyspark.ml.feature import PCA
pca = PCA(k=2, inputCol="features", outputCol="pcaFeatures")
model = pca.fit(output)
result = model.transform(output).select("pcaFeatures")
>>> result.show(5, truncate=False) +-----------------------------------------+ |pcaFeatures | +-----------------------------------------+ |[-2.8182395066394674,-5.6463498234127885]| |[-2.788223445314679,-5.149951351762905] | |[-2.6133745635497077,-5.182003150742132] | |[-2.7570222769675947,-5.0086535975757736]| |[-2.773648596054474,-5.65370708976261] | +-----------------------------------------+
Визуализация главных компонент
Исходный датасет состоял из 4 признаков. После применения PCA мы уменьшили их до двух главных компонент. Если 4 измерения представить сложно, то вот 2 запросто. Выполним визуализацию главных компонент с помощью Python-библиотеки Matplotlib. Но сначала нам нужно разбить столбец pcaFeatures на два признака, поскольку функция scatter принимает в виде аргументов данные по X и Y. В Spark это можно сделать с помощью VectorSlicer (о нем тут). Код для разделения исходного вектора на два признака в Spark:
from pyspark.ml.feature import VectorSlicer slicer = VectorSlicer(inputCol="pcaFeatures", outputCol="attr_1", indices=[0]) sliced = slicer.transform(result) slicer = VectorSlicer(inputCol="pcaFeatures", outputCol="attr_2", indices=[1]) sliced = slicer.transform(sliced)
>>> sliced.select('attr_1', 'attr_2').show(5)
+--------------------+--------------------+
| attr_1| attr_2|
+--------------------+--------------------+
|[-2.8182395066394...|[-5.6463498234127...|
|[-2.788223445314679]|[-5.149951351762905]|
|[-2.6133745635497...|[-5.182003150742132]|
|[-2.7570222769675...|[-5.0086535975757...|
|[-2.773648596054474]| [-5.65370708976261]|
+--------------------+--------------------+
Осталось только достроить диаграмму рассеяния. Чтобы использовать данные Spark-датафрейма, используется функция collect. Также выделим цветом вид ириса. Код для визуализация PCA и выделения данных Spark выглядит следующим образом:
import matplotlib.pyplot as plt
x = sliced.select('attr_1').collect()
y = sliced.select('attr_2').collect()
colors_map = [
0: '#b40426',
1: '#3b4cc0',
2: '#f2da0a',
]
# из кода выше Y = iris.target
colors = list(map(lambda p: colors_map[p], Y))
plt.scatter(x, y, c=colors)
plt.title(f'Visualizing the principal components')
plt.xlabel('Principal component 1')
plt.ylabel('Principal component 2')
plt.show()
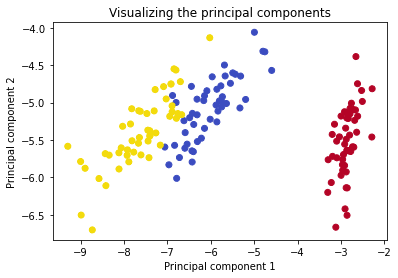
Полученный график хорошо демонстрирует результат уменьшения размерности. Посмотрите, как просто на полученных главных компонентах можно применить алгоритмы классификации или кластеризации.
Еще больше подробностей о подготовке датасетов в Spark, а также о методах уменьшения размерностей, включая PCA, на реальных примерах прикладных задач Big Data, вы узнаете на специализированном курсе «Машинное обучение в Apache Spark» в лицензированном учебном центре обучения и повышения квалификации разработчиков, менеджеров, архитекторов, инженеров, администраторов, Data Scientist’ов и аналитиков Big Data в Москве.



